본 게시글은 랜덤포레스트의 간략한 소개와 R 기반의 랜덤포레스트 모형 학습 과정을 소개한다.
전체적인 과정은 0. 랜덤포레스트 소개, 1. 패키지와 데이터 불러오기, 2. 데이터 분할과 학습, 3. 학습된 모형 평가, 4. 변수 중요도 순서이다.
0. 랜덤포레스트 (randomForest)
랜덤포레스트 사용하기 전, 꼭! 알아야 하는 랜덤포레스트의 특성을 소개하고 넘어간다.
이 특징을 이해해 두면, 코드 작성 과정에서의 이해나 결과 해석이 편해진다.
- 앙상블(Ensemble) 모형: Randomforest는 여러 개의 Decision Tree 모형을 조합하여 만든 앙상블 모형이다.
- 각 트리마다 무작위 추출 진행: 각각의 Decision Tree 학습에 사용되는 변수와 관측은 모두 무작위로 추출하여 사용한다.
Decision Tree를 조합하였다는 표현을 직관적으로 이해하기 위해, 아래와 같은 예시를 생각할 수 있다.
1번 Tree는 x<50일 때 y=9으로, 아닐 땐 y=4 예측한다. 2번 Tree는 x<30일 때 y=7로, 아닐 땐 y=5로 예측한다.
1번과 2번 Tree 모형을 평균내어, x<30 일 땐 (9+7)/2, 30<x<50 일 땐 (9+5)/2, 50<x일 땐 (4+5)/2로 예측한다고 이해하면 된다.
모형의 특징들로 인해, RandomForest는 다음과 같은 성징을 갖는다.
- 어느 데이터에서건 안정적인 성능 보장. (상대적으로)
- 특정 관측이나 변수에 대한 과적합 완화.
그렇기 때문에, 만약 제안하는 새로운 모형의 성능을 비교하거나 이 전에 알려지지 않은 새로운 문제에 도전할 때, RandomForest를 근거로 Baseline 성능을 확인하기도 한다.
1. 패키지와 와 데이터 불러오기.
randomForest 패키지를 설치한다.
(이후에 ranger라는 randomforest보다 더 빠른 학습 속도를 자랑하는 패키지가 개발되기도 했다. randomForest와 거의 사용법이 유사하다.)
##### Loading #####
install.packages("randomForest")
library(randomForest)
분석에 사용하고자 하는 데이터는 ISLR 홈페이지의 resources에서 다운받을 수 있다. (링크: https://www.statlearning.com/)
heart <- read.csv("./Heart.csv", row.names="X")
str(heart)
AHD는 Outcome 변수로 Heart Disease의 여부를 Yes/No로 나타낸다.
이 외의 변수는 연령(Age), 성별(Sex), 가슴 통증 정도(ChestPain), 각종 측정값 (RestBP, Chol, Fbs,...) 등으로 구성되어 있다.
2. 학습하기.
결측 값이 존재하나 본 게시글에서 다루지 않으므로 결측값을 생략하고 분석에 진행하였다.
각 데이터를 7:3의 비율로 학습과 평가용 데이터셋으로 구분한다.
그리고 학습용 데이터 셋을 이용해 RandomForest 학습 과정은 다음과 같다. [코드 확인!!]
각 학습에 주요하게 사용하는 옵션은 다음과 같다.
- ntree: 앙상블할 Tree 모형의 개수. 기본 값은 500개이지만, 관측이 적은 경우 트리의 수가 너무 많으면 과적합될 수 있으니, 적절히 줄여서 사용하자.
- mtry: 랜덤하게 뽑을 변수의 개수. 기본적으로 분류 문제에서 변수의 개수를 p로 두면, 분류 문제에서 $\sqrt{p}$ 회귀문제에서 $p/3$을 사용한다.
사용할 표본 선택에 대한 옵션 또한 제공하지만, 기본적으로 붓트스트랩(Bootstrap)에 근거하여 뽑고 있으니 건드리지 말자. (주어진 표본만큼 무작의 복원 추출) mtry도 기본 옵션을 사용하는 게 편하다.
# delete missing
heart.omit <- na.omit(heart)
# Sample Size 303 -> 297
n <- nrow(heart.omit)
idx <- sample.int(n)
##### split dataset #####
tr_n <- round(n*0.7)-1
test_n <- n-tr_n
tr_idx <- idx[1:tr_n]
test_idx <- setdiff(1:n, tr_idx)
train_y <- as.factor( heart.omit$AHD[tr_idx] )
test_y <- as.factor( heart.omit$AHD[test_idx] )
heart.omit$AHD <- NULL
train_df <- heart.omit[tr_idx,]
test_df <- heart.omit[test_idx,]
###### train rf #####
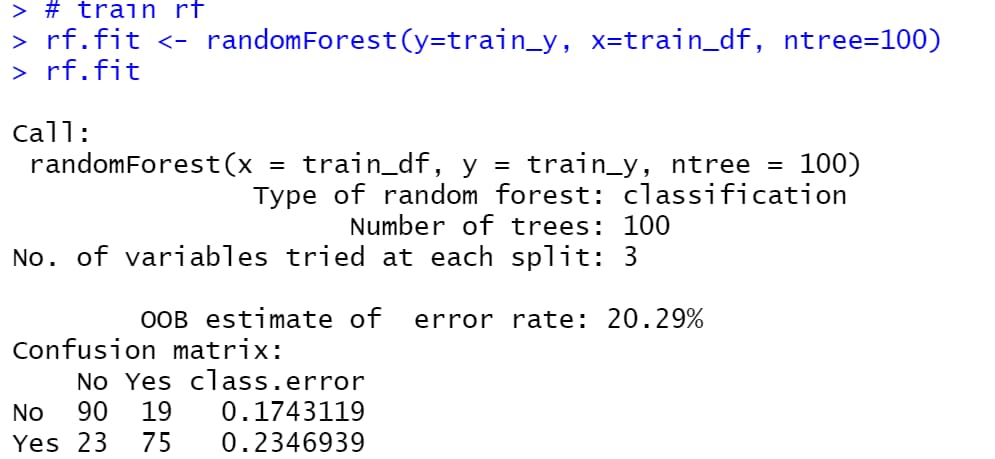
rf.fit <- randomForest(y=train_y, x=train_df, ntree=100)
rf.fit
랜덤포레스트 학습된 결과를 해석해보자.
OOB estimate of error rate는 Out-of-bag error의 추정량을 의미한다.
앞에서 RandomForest는 하나의 트리마다 무작위 표본과 무작위 변수를 선택하는 과정이 있다고 이야기했다.
여기서 선택된 무작위의 표본을 내 가방(Bag)에 넣었다고 표현하고, 내 가방 밖에 있는, 즉 선택되지 않은 표본을 Out-of-Bag이라고 표현한다.
즉 선택된 무작위 표본과 무작위 변수로 학습된 하나의 트리 모형이 선택되지 않은 표본에서도 성능이 좋은지를 평가한 것이다.
3. 모형 평가하기.
ntree=100으로 설정한 모형은 계산해 보면 대략적으로 정확도가 0.86이다.
predict 함수는 인풋에 따라 예측할 값의 type의 디폴트 옵션이 바뀌는데 randomForest 기반의 분류 문제에선 기본적으로 type=class옵션이 사용된다.
하지만 type=prob을 사용하여 각 클래스에 속할 확률을 계산할 수도 있고, cut-off 기준이 0.5가 아니라 0.4로 사용하고 싶다면 각 클래스에 속할 확률 값을 출력하여, 직접 분류할 수 도 있다.
###### evaluate the model ######
rf.fit100 <- randomForest(y=train_y, x=train_df, ntree=100)
fit100_class <- predict(rf.fit100, newdata=test_df, type="class")
cm <- table("predicted"=fit100_class, "true"=test_y)
fit100_acc <- (cm[1,1]+cm[2,2])/sum(cm)
fit100_acc # 0.8555556
fit100_prob <- predict(rf.fit100, newdata=test_df, type="prob")
head(fit100_prob)
# No Yes
# 5 1.00 0.00
# 8 0.50 0.50
# 11 0.68 0.32
# 13 0.34 0.66
# 16 0.86 0.14
# 18 0.78 0.22
일반적으로 랜덤포레스트가 과적합을 완화한다고 알려져 있지만, 그렇다고 완전히 과적합의 문제에서 자유로운 것은 아니다.
항상 그런 것은 아니지만, ntree가 너무 많으면 과적합이 발생할 수 있다.
분석에 사용한 데이터의 크기가 297이라, 디폴트 옵션인 ntree=500보다 ntree=100일 때 성능이 더 좋은 것을 확인할 수 있다.
rf.fit500 <- randomForest(y=train_y, x=train_df, ntree=500)
fit500_class <- predict(rf.fit500, newdata=test_df, type="class")
cm <- table("predicted"=fit500_class, "true"=test_y)
fit500_acc <- (cm[1,1]+cm[2,2])/sum(cm)
fit500_acc # 0.84444444. 변수 중요도 확인.
적합된 randomForest 안에서 변수 중요도를 대략적으로 파악할 수 있도록 importance를 제공한다.
가장 기본으로 사용되는 값은 MeanDecreaseGini인데, 해당 변수를 기준으로 분류한 뒤의 얼마나 불순도가 낮아졌는지를 의미한다.
위 기준을 근거로, 변수 중요도가 높은 순서대로 정렬하여 확인할 수 있다.
###### variable importance ######
ord_name <- rownames(rf.fit100$importance)[order(rf.fit100$importance, decreasing=TRUE)]
rf.fit100$importance[ord_name,]

'R Programming > Analysis' 카테고리의 다른 글
| [R] shapviz 패키지로 SHAP Value 구하고 해석하기 (0) | 2024.08.22 |
|---|---|
| [R] Auto Correlation 데이터 생성과 Durbin-Watson 검정 (1) | 2023.12.28 |
| [R] Cox 분석을 위한 생존 시간 데이터 생성 (시뮬레이션 코드) (2) | 2023.12.22 |
| [R] 부트스트랩 신뢰 구간 (Bootstrap Confidence Intervals) 계산 (2) | 2023.11.27 |
| [R] 몬테카를로 실험 기반의 검정 (monte carlo test) (2) | 2023.10.11 |


